Récemment, une équipe de plus de 200 chercheurs annonçait l’achèvement du séquençage du génome du rat de laboratoire, le Rattus norvegicus. La nouvelle faisait la une du numéro du 1er avril de Nature, et il ne s’agissait pas d’un poisson d’avril.
Parmi les cosignataires de cet article se trouve un chercheur de l’Université, Guillaume Bourque, stagiaire postdoctoral au Centre de recherches mathématiques. M. Bourque est plus précisément le concepteur d’un algorithme qui permet de comparer entre elles des sections de chromosomes d’espèces différentes.
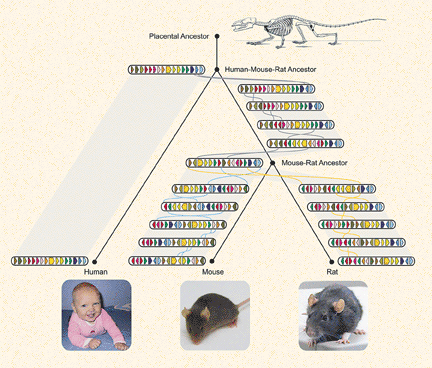
Après la souris et l’être humain, le rat est le troisième mammifère dont le séquençage du génome est maintenant terminé. La connaissance de ces trois génomes permet, grâce à l’outil élaboré par Guillaume Bourque, de reconstituer en partie le profil génétique de l’ancêtre de tous les mammifères, soit un rongeur à fourrure qui a vécu il y a près de 90 millions d’années. Le chercheur et ses deux collaborateurs – les professeurs Pavel Pevzner et Glenn Tesler, de l’Université de Californie à San Diego – ont de façon encore plus précise recréé le portrait génétique de l’ancêtre commun à la souris et au rat.
 |
|
Guillaume Bourque |
«Notre algorithme permet de repérer des blocs d’homologie dans les génomes de diverses espèces et d’observer de quelle manière ces blocs ont été réarrangés au cours de l’évolution», explique Guillaume Bourque.
Pour nous aider à mieux comprendre ce dont il s’agit, le chercheur emploie l’analogie d’un jeu de cartes que l’évolution aurait brassé différemment pour chacune des trois espèces: grâce à l’algorithme, à partir des trois brassages observés, il est possible de reconstituer l’ordre des cartes du jeu initial, ce qui correspond à l’ancêtre des mammifères. L’algorithme permet également de repérer les cartes qui sont restées au même endroit et de les distinguer de celles qui ont été déplacées. Le brassage des cartes correspond aux réarrangements génomiques désignant les mutations qui modifient l’ordre des gènes ou les échanges de gènes entre les chromosomes.
90 % de gènes en commun
Les données avec lesquelles l’algorithme doit opérer ne sont toutefois pas du même ordre de grandeur que celui d’un paquet de cartes: ce ne sont pas 52 éléments d’information qu’il faut analyser mais plusieurs milliards. Le génome de l’être humain contient en effet près de 3 milliards de nucléotides, celui du rat 2,7 milliards et celui de la souris 2,6 milliards.
L’outil de Guillaume Bourque et de ses collaborateurs a permis de repérer 280 blocs d’homologie distribués dans l’ensemble des chromosomes. Les résultats ont montré que les trois génomes présentent 40 % de bases nucléotides ayant une homologie très élevée. Étant donné que la majorité des gènes se trouve dans ces séquences communes du génome, l’être humain, le rat et la souris ont ainsi plus de 90 % de gènes en commun.
Les chercheurs ont plus précisément fait porter leurs travaux sur le chromosome X parce que celui-ci échange très peu de matériel génétique avec les autres chromosomes. L’étude a réussi à démontrer que la configuration des blocs d’homologie sur ce chromosome est probablement la même chez l’homme que chez l’ancêtre des mammifères.
Plus humain que rongeur
|
Illustration de la couverture de Genome Research du mois d’avril conçue par Guillaume Bourque. On y voit les 16 blocs d’homologie (représentés par des couleurs différentes) retrouvés sur le chromosome X de l’être humain, du rat et de la souris, tels qu’ils ont été réarrangés au fil de l’évolution et qui permettent d’établir le profil possible de ce chromosome chez un ancêtre commun aux trois espèces. |
| |
«Ce qui est étonnant, c’est qu’à certains niveaux ces trois espèces présentent la même distance génétique l’une par rapport à l’autre, affirme M. Bourque. Le rat est aussi éloigné de la souris qu’il l’est de l’être humain.»
Autrement dit, l’être humain est aussi près du rat que celui-ci l’est de la souris! Nous serions d’ailleurs plus près de notre ancêtre commun que le sont le rat et la souris même si cet ancêtre était un petit rongeur.
L’énorme différence entre l’être humain et le rat serait en partie attribuable à l’emplacement des blocs d’homologie sur les chromosomes, estime le chercheur. «L’emplacement des séquences de nucléotides a un effet activant ou désactivant sur les gènes, explique-t-il. Les réaménagements de ces blocs peuvent donc avoir un effet considérable ou négligeable selon l’endroit où ils se retrouvent.»
Le nombre de réarrangements qui ont influé sur chacune de ces trois espèces est comparable, soit de l’ordre d’une cinquantaine. Mais comme ils sont survenus sur une période plus courte chez les rongeurs que chez les primates (90 millions d’années nous séparent de l’ancêtre commun aux trois contre 12 à 24 millions d’années qui séparent le rat et la souris de leur propre ancêtre), cela signifie que les mutations ont été plus fréquentes dans la lignée des rongeurs que dans celle des primates.
Aux yeux des signataires de l’article de Nature, le haut pourcentage de gènes partagés entre l’être humain et le rat confirme que cet animal constitue un bon modèle pour l’étude des maladies d’origine génétique chez l’homme. Toutefois, une bonne part des 10 % de gènes que partagent la souris et le rat et qui sont absents chez l’humain seraient associés à l’odorat et à l’élimination des agents toxiques. Le rat aurait donc plus de gènes liés à la désintoxication, ce qui appelle à la prudence dans le recours à cet animal pour évaluer la toxicité d’un médicament.
Outre le dossier de Nature, les résultats des travaux de Guillaume Bourque, Pavel Pevzner et Glenn Tesler sont publiés dans l’édition spéciale d’avril de la revue Genome Research, qui consacre son numéro aux recherches sur les trois génomes. La mise au point de l’algorithme découle notamment d’études antérieures effectuées par David Sankoff quand il était professeur au Centre de recherches mathématiques. Cette recherche de l’architecture du génome ancestral avait alors reçu le nom de «problème de synténie».
Daniel Baril